Sequencing DNA

Sekuensing DNA atau pengurutan basa DNA adalah penemuan urutan basa nitrogen (adenin, guanin, sitosin, dan timin) pada sampel DNA. Sekuensing berguna untuk bioteknologi, biologi molekuler, dan genomika. Tahun 1970 merupakan awal pengembangan sekuensing DNA dengan metode kromatografi. Selanjutnya diperkenalkan metode sekuensing DNA dengan menggunakan metode dye-based sequencing Pada awalnya alat untuk membaca urutan basa DNA hanya mampu menentukan urutan basa DNA dengan panjangnya kurang dari 1.000 basa. Oleh sebab itu, dalam sekuensing genom, terlebih dahulu molekul DNA dipotong menjadi ukuran yang lebih kecil agar dapat dibaca dengan alat tersebut. Hasil data urutan DNA kemudian disusun ulang menggunakan program komputer sehingga urutan keseluruhan DNA dalam inti sel dapat diketahui (Franca et al.2002).
Teknologi baru diperlukan untuk mengakselerasi pemanfaatan koleksi sumber daya genetik pada Bank Gen untuk program pemuliaan tanaman. Hal ini dapat dilakukan dengan memperbaiki metode genotyping dan phenotyping serta meningkatkan akses untuk mendapatkan materi genetik yang disimpan pada Bank Gen. Sebelum ditemukannya alat yang mampu membaca urutan DNA secara paralel dalam jumlah besar, perunutan basa-basa DNA (sequencing) memakan waktu, tenaga, dan biaya yang sangat besar (Kling 2005).
Perkembangan teknologi next-generation sequencing (NGS) mengakibatkan revolusi di dalam bidang bioteknologi tanaman, terutama untuk menyediakan data sekuensing yang lebih besar daripada metode sekuensing Sanger. Waktu yang diperlukan untuk mendapatkan data yang sama dapat dipangkas menjadi hanya beberapa bulan serta penurunan biaya sekuensing genom dapat mengatasi masalah dalam genotyping tersebut. Teknologi NGS mempercepat pemetaan sekuen genom acuan (reference genome sequence) dari berbagai spesies tanaman, dari tanaman model sampai tanaman budi daya yang bernilai ekonomi tinggi, dari tanaman model Arabidopsis thaliana sampai tanaman yang berukuran genom besar dan kompleks seperti jagung, kelapa sawit, kedelai, tebu, dan gandum. Sampai saat ini, setidaknya 39 spesies tanaman telah tersedia peta genom acuannya (Dhanapal 2012; Van et al. 2013).
Metode sekuensing DNA yang pertama kali digunakan adalah metode sekuensing Maxam-Gilbert merupakan metode sekuensing DNA pertama yang memanfaatkan bahan kimia untuk separasi dan penandaan urutan basa. Metode ini memanfaatkan fosfat radioaktif untuk melabeli ujung 3’ DNA yang akan disekuensing. Fragmen DNA berlabel di ujung 3’ dikenai pembelahan acak pada posisi adenin, timin, guanin, dan sitosin dengan memanfaatkan agen kimia tertentu. Reaksi kimia terhadap komponen basa didasari oleh tiga komponen kunci, yakni modifikasi basa, penghilangan basa yang telah dimodifikasi dari gula, dan pemutusan strand DNA pada posisi gulanya. Empat reaksi produk lalu diseparsi dengan elektroporesis gel poliakrilamit. Sekuen akan mudah untuk dibaca ada empat lajur gel.
Setelah metode sekuensing Maxam-Gilbert, ditemukan metode Sanger (Sanger dideoxy sequencing). Sekuensing Sanger terkenal karena akurasi dan panjang pembacaannya (800–1.000 bp) telah mendominasi selama hampir 30 tahun sebagai metode baku untuk analisis genotipe. Metode ini menggunakan DNA templat dan memerlukan primer spesifik untuk reaksi sekuensing (Sanger et al. 1997). Keterbatasan dalam sekuensing Sanger dapat diatasi dengan metode NGS, antara lain NGS dapat menganalisis urutan dari satu kromosom, cost efective untuk sampel dalam jumlah besar, dan juga high-throuhgput. Tulisan ini bertujuan untuk mengulas perbedaan beberapa pemanfaatan teknologi sekuensing DNA secara efisien dan efektif untuk menunjang program pemuliaan tanaman.
METODE SEKUENSING
Metode Maxam Gilbert
Metode sekuensing Maxam-Gilbert merupakan metode sekuensing DNA pertama yang memanfaatkan bahan kimia untuk separasi dan penandaan urutan basa. Metode ini memanfaatkan fosfat radioaktif untuk melabeli ujung 3’ DNA yang akan disekuensing. Fragmen DNA berlabel di ujung 3’ dikenai pembelahan acak pada posisi adenin, timin, guanin, dan sitosin dengan memanfaatkan agen kimia tertentu. Reaksi kimia terhadap komponen basa didasari oleh tiga komponen kunci, yakni modifikasi basa, penghilangan basa yang telah dimodifikasi dari gula, dan pemutusan strand DNA pada posisi gulanya. Empat reaksi produk lalu diseparsi dengan elektroporesis gel poliakrilamit. Sekuen akan mudah untuk dibaca ada empat lajur gel.
Prinsip metode ini yaitu separasi basa berdasarkan struktur kimia. Penggunaan bahan kimia untuk degradasi berdasarkan pada struktur setiap basa. Setiap basa didegradasi secara spesifik sehingga dapat menampilkan pita spesifik pada setiap urutan dalam gel poliakrilamit. Setiap reaksi seragam pada sekuen yang sama. Proses detail dalam metode Maxam-Gilbert yaitu separasi dengan bahan kimia, sintesis pertukaran [γ-32P] ATP, pelabelan pada ujung 5’, pelabelan pada ujung 3’, separasi stran, perlakuan gel elution, metilasi purin, pemisahan guanin dan adenin, pemisahan timin dan sitosin dan visualisasi.
Bahan kimia yang digunakan dalam separasi yaitu dimetil sulfa. Senyawa ini berfungsi memitilasi guanin pada DNA posisi N7 dan adenin pada N3. Ikatan glycosidic pada methylated purine tidak stabil dan mudah dirusak oleh pemanasan pada pH netral sehingga menghasilkan gula yang bebas. Pelakuan dengan 0.1 M alkali pada 90 oC dapat memisahkan gula dari fosfat. Ketika proses telah menghasilkan fragmen DNA berlabel pada poliakrilamit, autoradiograph akan memancarkan pita gelap dan pita tipis. Pita gelap muncul dari menghancuran guanin yang termetilasi 5-fold lebih cepat dibanding adenin. Pola Strong guanin/weak adenine mengandung hampir setengah informasi sekuensing. Untuk menentukan urutan basa, Maxam & Gilbert (1977) melakukan pembandingan informasi yang terkandung pada kolom yang berbeda.
Peningkatan pemisahan adenin melalui ikatan glycosidic methylated adenosine kurang stabil dibanding dari ikatan methylated guanosianine. Pemisahan dilakukan dengan alkali sehingga menghasilkan pita gelap dan tipis. Pita gelap menandakan adenin, pita tipis menandakan guanin. Pemisahan sitosin dan timin melalui hidrazine bereaksi dengan timin dan sitosin, yang mana bersifat destruktif dan dapat meninggalkan bosylurea. Reaksi lanjut dari dari hydrazine dapat menghasilkan hydrazone. Setelah, setengah proses hydrazinolysis pada 15018 M hidrazine cair pada 20 oC. DNA dipisah pada 0.5 M piperidin. Siklus secondary amine, sebagai bebas basa, menggantukan semua produk reaksi hydrazine dari gula dan katalis β-elimination pada fosfor. Penampakan final mengandung pita yang sama antara sitosin dan timin.
Sintesis pertukaran [γ-32P] ATP menggunakan dialyze glyceraldehyde-3-phosphate dehydrogenase yang mengandung amonium sulfat dengan Tris.HCl, mercaptothanol, EDTA, dialyze 3-phosphoglycerate kinase dengan larutan NAD+. Dialyzed dehydrogenase dikombinasi dengan dialyzed kinase, sedimen dilarutkan ulang pada pellet lalu didestilasi oleh air dua kali untuk menghilangkan ammonium sulfat. Setelah itu digunakan EDTA untuk mengurangi glutathionine dan garam setelah penambahan larutan destilat. Reaksi dilanjutkan dengan mengikuti reaksi charmography pada PEI Selulosa. Proses setelahnya adalah dengan penambahan EDTA dan pemanasan untuk menonaktifkan enzim, yang dilanjutkan dengan penambahan etanol sebelum disimpan pada minus -20 oC.
Peroses pelabelan dimulai dengan pelabelan pada ujung 5’. Fosforilasi ujung 5’ meliputi denaturasi dengan spermidine untuk meningkatkan hasil 15-fold dengan fragmen flush-end. Pada prosesnya, dilakukan pencampuran DNA terfosforilasi, glisin NaOH, spermidine yang kemudian dipanaskan dan didinginkan. Selanjutnya dilakukan penambahan glisine NaOH, MgCl2, dithiothreitol, [γ-32P] ATP dan beberapa unit polinukleatida kinase. Pemanasan dan penambahan amonium asetat, tRNA dan etanol, lalu dicampurkan dan didinginkan. Sentrifugasi dilakukan pada 12.000x g untuk menghilangkan supernatan lalu dikeringkan.
Pelabelan pada ujung 3’ dilakukan dengan proses [α-32P] ATP dan terminal transferase digunakan untuk adenylate. Larutkan DNA dengan Tris.HCl, EDTA dilakukan pemanasan dan pendinginan. selanjuttnya dilakukan penambahan sodium cacodylate, CoCl2, lalu dicampur dithiothereitol, [α-32P] ATP, dan beberapa unit terminal transferase. Pemanasan dan penambahan amonium asetat, tRNA, etanol untuk kemudian dipresipitasi, sentrifugasi, dibersihkan dan dikeringkan. Larutkan pellet yang diperoleh dicampur EDTA lalu dipanaskan dan ditambahkan glyserol untuk pewarnaan juga untuk pemisahan strand.
Pemisahan utas stran basa menggunkan larutkan DNA dengan NaOH, glyserol, EDTA, xylene cyanol/, bromphenol biru. Bahan dimasukkan pada poliakrilamit, bisacrylamit, Tris.borate, EDTA dan elektroporesis. Gel yang digunakan adalah gel yang tebal dengan slot yang luas. Elusin gel dilakukan dengan penghalusan gel agar dapat dicampur dan dituangkan bersama siliconized 5-mm glass rod, penambahan amonium asetat, magnesium asetat, sodium dedocyl dan EDTA, kemudian ditutup dengan parafin.
Selanjutya dilakukan metilasi purin sebagian. Proses ini dilakuakn dengan penggabungan sonicated DNA yang membawa DNA dengan 32P-end-labeled DNA, sodium cocodylate, MgCl2, EDTA, dicampur, didinginkan lalu diberi penambahan metyl sulfat dan pemanasan. pemberhentian reaksi dilakuakn dengan penambahan larutan stop dan tRNA, lalu ditambahkan dengan 750 etanol, didinginkan lalu spin dan presipitasikan. Pemisahan guanin melalui proses DNA termetilasi dilarutkan pada sodium pospat, EDTA, dan koleksi cairan yang terdapat pada bawah tube dengan pemutaran. Setelah dipanaskan dan dispin dilakukan penambahan NaOH dan urea-dye.
Pemisahan strong guanine/weak adenin dilakukan dengan DNA termetilasi dilarutkan pada sodium pospat, EDTA, dan koleksi cairan yang terdapat pada bawah tube dengan pemutaran. Setelah dipanaskan dan dispin di tambahkan NaOH dan urea-dye. Selannjutnya dilakukan strong adenin/weak guanin dengan mengguakan DNA termetilasi dilarutkan pada air destilasi. Didinginkan lalu ditambahkan HCl dan didingankan. Setelah dua hari, dilakukan penambahan sodium asetat dan etanol. Larutkan dengan NaOH, EDTA dan panaskan lalu tambahkan urea-dye. Pemisahan alternatif guanin menggunakan DNA termetilasi dilarutkan dengan piperidin. Reaksi membuka 7-MeG ikatan glycosidic, lalu dipindahkan pada ring-opned dari gula, mengeliminasi pada kedua fosfat untuk memisahkan DNA dimanapun G termetilasi. Lalu larutkan residu, NaOH, EDTA dan disiapkan pada gel.
Pemisahan alternatif strong adenin/weak sitosin menggunakan kombinasi NaOH, EDTA dengan DNA dan 32P-end label DNA. Strong alkali membuka adenin dan cincin sitosin. Lalu membuka cincin dan digantikan dengan fosfat dan mengeliminasi dengan piperidin. Larutkan pellet dengan piperidin. Selanjutnya lyophilize dua kali yang ditambahkan urea-dye. Pemisahan timin dan sitosin menggunakan kombinasi dengan air destilat, DNA, dan 32P end-labeled DNA. Penambahan hidrazine dilakukan dan selanjutnya dipisah pada piperidin. Siklus secondary amine, sebagai bebas basa, menggantukan semua produk reaksi hydrazine dari gula dan katalis β-limination pada fosfor. Pemisahan timin dilakukan dengan penggantian air pada reaksi hydrazinolysis dengan NaCl, lalu tambahkan pirimidin agar muncul pita timin. Waktu reaksi bersifat stabil dan sama dari satu reaksi ke reaksi lainnya. Produk yang dihasilkan sebanyak 1-100 basa dalam satu kali reaksi. Contoh hasil sekuensing metode Maxam-Gilbert telihat pada Gambar 1.
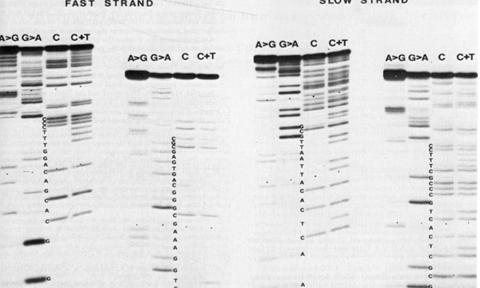
Gambar 1 Contoh hasil sekuensing metode Maxam-Gilbert (Sumber: Maxam & Gilbert (1977))
Teknik Medode Sanger
Metode sanger menggunaan berbagai komponen yaitu DNA utas tunggal dalam sebagai template DNA, primer yang sesuai (sepotong kecil DNA yang berpasangan dengan template DNA yang berfungsi sebagai starting point untuk replikasi, DNA polymerase (enzim yang mengkopi DNA selanjutnya menambahkan nukleotida baru ke ujung 3 dari template), nukeotida normal dan dideoxynucleotide yang dilabel (radioaktif atau dengan pewarna fluorescent (Sangger 1997).
Tahapan metode sekuensing Sanger yang pertama adalah menyediakan dsDNA (double strand DNA), dsDNA (double strand DNA) dipotong menjadi ssDNA (single strand DNA), ssDNA hasil potongan dari dsDNA sebelumya dijadikan sebagai template (cetakan) DNA. Bahan yang digunakan untuk sekuensing adalah template DNA sebagai cetakan, primer sebagai landasan yang mengenal situs spesifik pada DNA template untuk memulai proses polymerase, dNTP (deoksi nukleotida tri phospat) sebagai sumber nukleotida pada proses polymerase sekuensing yang terdiri atas 5 jenis, yaitu dATP (deoksi adenin tri phospat), dGTP (deoksi guanin tri phospat), dUTP (deoksi urasil tri phospat), dCTP (deoksi citosin tri phospat), dan dTTP (deoksi timin tri phospat). ddNTP (dideoksi nukleotida tri phospat) berfungsi untuk menghentikan/terminasi proses enzim polymerase, sehingga proses perbanyakan nukleotida terhenti. Enzim polymerase berfungsi untuk reaksi polimerisasi atau perbanyakan nukleotida. Kemudian masing-masing tabung reaksi diberikan ddNTP, yaitu ddGTP, ddCTP, ddATP, dan ddTTP selannjutnya masing-masing tabung diisi dengan dNTP, sebagai sumber nukleotida pada proses polimerasi yaitu dGTP, dCTP, dATP, dan dTTP. dNTP dimasukkakn ke dalam tabung reaksi ditambahkan primer ke dalam tabung reaksi. Primer berfungsi mengenali situs spesifik pada DNA template, juga berfungsi sebagai landasan/pijakan untuk memulai polimerisasi. Kemudian ditambahkan enzim polimerase (taq-polymerase) untuk memulai proses polimerisasi.
Enzim polymerase mengkatalisis pembentukan polinukleotida dari nukleotida dNTP (deoksi nukleotida tri phospat), enzim taq-polymerase mengkatalisis pembentukan ikatan antara nukleotida, deoksi-nukleotida (ddNTP) akan berikatan dengan polimer nukleotida dengan adanya ddNTP (deoksinukleotida) mengakibatkan terhentinya/terminasi proses polimerase, sehingga dihasilkan rantai polinukleotida yang berbeda panjangnya. Penggunaan ddNTP menghasilkan beberapa rantai polinukleotida berbeda, kemudian dari tabung reaksi tersebut dipersiapkan untuk di alirkan pada gel agarosa yang mengakibatkan perbedaan letak pada gel agarosa. Polinukleotida yang paling pendek bermigrasi/pergerakannya paling cepat pada gel agarosa. Hasil pembacaan sekeuensing dari arah 5’ ke 3’ adalah rantai kompemen, yaitu 5’ AGCCGATCC 3’. Sehingga DNA templatenya adalah 5’ GGATCGGCT 3’
Next Generation Sequencing (NGS)
Generasi kedua teknologi sekuensing setelah generasi pertama Sanger dikenal dengan next generation sequencing (NGS). Istilah NGS secara kolektif digunakan untuk mendeskripsikan semua teknologi sekuensing selain teknologi sekuensing Sanger. Teknologi NGS membaca templat DNA secara acak (random) sepanjang seluruh genom dengan membuat potongan-potongan pendek DNA genom, kemudian menyambungkannya dengan adapter (potongan DNA pendek yang didesain khusus untuk tujuan ini) agar dapat dibaca oleh mesin NGS secara random selama proses sintesis DNA. Dengan demikian, teknologi NGS sering disebut dengan sekuensing paralel secara masif. Panjang bacaan sekuen DNA yang dihasilkan mesin NGS jauh lebih pendek dibandingkan bila menggunakan mesin sekuensing dengan metode Sanger (Paterson 2010).
NGS menghasilkan panjang sekuen DNA antara 50-500 bp. Karena sekuen yang dihasilkan NGS pendek, sekuensing setiap fragmen DNA mesti dilakukan lebih dari sekali ukuran genom (genome sequence coverage). Sebagai contoh, bila sequence coverage suatu genom 30 kali berarti fragmen-fragmen DNA pada genom tersebut disekuen 30 kali sehingga setiap fragmen DNA dalam genom dibaca mesin 30 kali. Hal ini dilakukan untuk menjaga akurasi data hasil sekuensing.
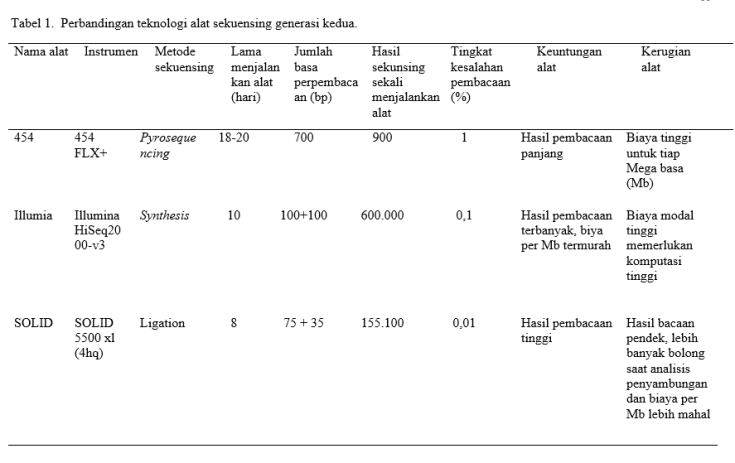
Penerapan inovasi teknologi NGS sangatlah cepat sama sekarang. Teknologi NGS yang bekembang sekarang dibagi tiga yaitu Roche/454 pyrosequencing platform, Illumina Solexa polymerase sequencing platform, dan ABI/SOLID ligase sequencing technology. Dibandingkan dengan metode sekuensing Sanger, ketiga teknologi NGS ini menghasilkan data sekuen yang jauh lebih banyak dalam sekali menjalankan alat. Dengan demikian, alat ini dikenal dengan high throughput sequencing platforms (Ansorge 2009). Prinsip dasar ketiga alat NGS tersebut berbeda, baik dalam menghasilkan data sekuen, kualitas data yang dihasilkan maupun biaya sekuensing (Tabel 1). Alat Roche/454 menghasilkan data sekuen terpanjang, tetapi kuantitas data sekuennya terendah (lowest throughput). Jumlah sekuen basa yang dihasilkan Illumina/Solexa tertinggi (highest throughput), tetapi panjang bacaannya hanya sekitar 100 basa. Panjang bacaan yang dihasilkan ABI/SOLID terpendek, hanya sekitar 50 basa, tetapi tingkat kesalahan pembacaan DNA pada proses sekuensing (error rate) paling rendah.
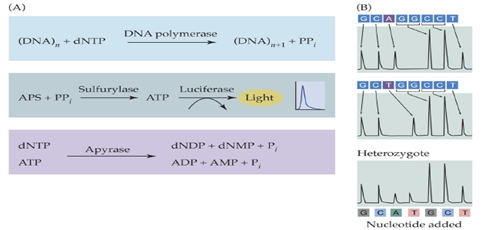
Teknologi NGS merupakan revolusi metode sekuensing dan menghasilkan data sekuen yang luar biasa besar dalam waktu relatif singkat dibandingkan dengan metode sekuensing Sanger. Oleh karena itu, diperlukan sumber daya teknologi informasi dan ahli bioinformatika untuk menganalisis data yang dihasilkan sehingga bermanfaat untuk mendukung program pemuliaan tanaman bernilai ekonomi tinggi. NGS menggunakan dua macam metode yaitu pertama dengan pencaran cahaya – pyrosequencing seperti 454 sekuensing (Gambar 1)
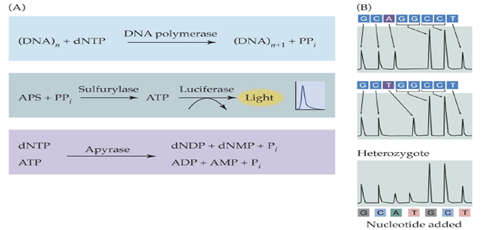
Gambar 1 Sekunsing dengan light emission – Pyrosequencing
Metode kedua yang dapat digunakan dalam NGS yaitu sekunsing permukaan (sequencing on surfaces) – polony sequencing. Metoni ini menguanakan koloni PCR pada permukaan (poloni), penggunaan matriks akrilamida untuk separasi utas basa-basa nukleotida, penggunaan DNA sangat encer, siklus penggabungan nukleotida fluoresen dan pembacaan sekitar 20-25 nukleotida per poloni (Gambar 2).
Gambar 2 sekunsing permukaan (sequencing on surfaces) – polony sequencing.
Pyrosekunsing adalah tenik pemetaan DNA berdasarkan deteksi pirofosfat (PPi) yang dilepaskan selama sintesis DNA. Teknik ini memanfaatkan reaksi enzimatik yang dikatalisis oleh ATP sulfurilase dan lucifelirase untuk pirofosfat inorganik yang dilepaskan selama penambahan nukleotida. Perkembangan teknik ini dimulai pada 1990 oleh Mostafa Ronaghi dari Royal Institute of Technology, Stockholm.
Prinsip metode sekuensing DNA ini bekerja berdasarkan urutan nuklotida dalam DNA dengan menggunakan prinsip sekuensi dari sintesis DNA. Teknik pyrosequencing menggunakan DNA yang diimobilisasi dalam fase solid dan diberi perlakuan dengan sistemenzim. Teknik ini dilakukan dengan penambahan nukleotida yang berbeda secara berurutan kemudian cahaya yang dihasilkan dapat dideteksi untuk mengetahui di mana letak nukleotida dengan jenis tersebut berada, dilanjutkan dengan penambahan nukleotida berikutnya dan terus berulang-ulang hingga seluruh sekuen DNA terpetakan. Oleh karena itu pada teknik ini diperlukan proses pencucian di antara penambahan basa nukleotida untuk menghilangkan nukleotida pada proses sebelumnya. Untaian tunggal yang akan disekuensing akan mensintesis untai komplemennya dengan enzim DNA polimerase dalam proses sintesis untai komplemen dideteksi dengan menggunakan enzim kemiluminesens, di mana akan dihasilkan cahaya ketika penambahan nukleotida ke DNA template dan cahaya tersebut yang akan diinterpretasikan sebagai sekuen hasil. Mekanisme pyrosekunsing terlihat pada Gambar 3 (Fakruddin 2013).
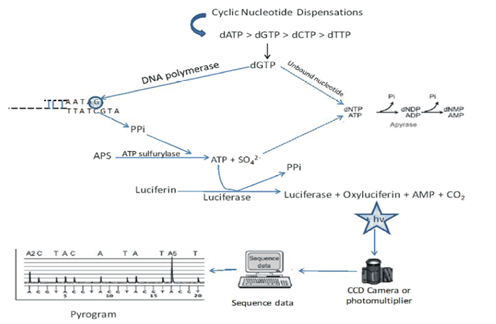
Gambar 3 Skema Pyrosequencing
Teknologi sekuensing illumina menggunakan metode sekuensing sintesis dimulai sejak 2010. Teknik ini berkerja dalam tiga proses dasar: memperbanyak sekuen/amplifikasi, pengurutan, dan menganalisis. Prosesnya dimulai dengan DNA yang dimurnikan. DNA akan dipotong kecil-kecil dan diberi adaptor, indeks dan jenis modifikasi molekuler lainnya yang bertindak sebagai titik referensi selama amplifikasi, sekuensing, dan analisis. DNA yang dimodifikasi dimuat ke chip khusus di mana amplifikasi dan sekuensing akan berlangsung. Sepanjang bagian bawah chip adalah ratusan ribu oligonukleotida (potongan DNA sintetis pendek).
Oligonukleotida akan berintraksi dengan fragmen DNA yang memiliki urutan komplementer. Jika terjadi penempelan oligonukleotida dengan DNA maka akan terjadi proses klasterilisasi. Selanjutnya, primer dan nukleotida yang dimodifikasi masuk ke chip. Nukleotida-nukleotida ini memiliki blokade reversibel 3′ yang memaksa primer untuk menambahkan hanya satu nukleotida pada suatu waktu dan juga tag fluoresen. Setelah setiap putaran sintesis, kamera mengambil gambar chip. Komputer menentukan basis apa yang ditambahkan oleh panjang gelombang dari tag fluoresensi dan mencatatnya untuk setiap titik pada chip. Setelah setiap putaran, molekul-molekul yang tidak dimasukkan dicuci. Proses ini berlanjut hingga molekul DNA lengkap diurutkan. Proses ilumina sekuensing terlihat pada Gambar 4-6 (Ansorge 2009).
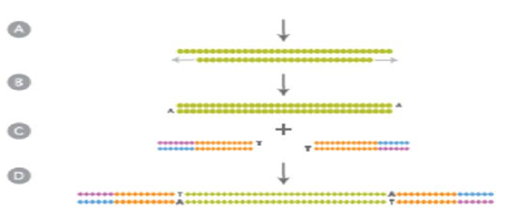
Gambar 4 Proses illumina sekuensing. (A). DNA fragmen, (B). Memperbaiki ujung dan menambah A, (C). Adaptor ligase dan (D). Penentuan DNA ligase
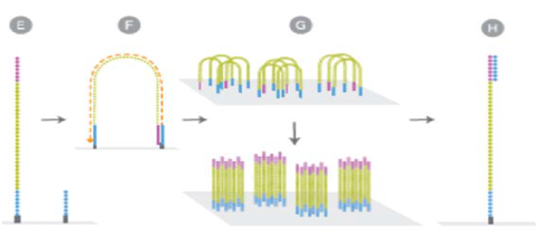
Gambar 5 Proses illumina sekuensing. (E). Pelekatan ujung DNA, (F). Amplifiaksi, (G). Hasil klaster dan (H). Penempelan primer
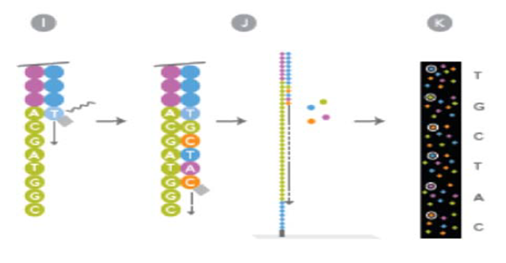 Gambar 6 Proses illumina sekuensing. (I). Perpanjangan (J). Pengulanggan langkah (I) untuk pembacaan dan (K). Hasil akhir urutan sekuen
Gambar 6 Proses illumina sekuensing. (I). Perpanjangan (J). Pengulanggan langkah (I) untuk pembacaan dan (K). Hasil akhir urutan sekuen
SOLiD sekunsing atau sering disebut Sequencing by Oligonucleotide Ligation and Detection (SOLiD) adalah metode sekuensing yang telah tersedia sejak tahun 2006. Metode ini menggunkan metode sekuensing ligasi dan dilakukan dengan persiapan template dari emulsi PCR. Metode ini memiliki kesamaan dengan metode pyrosekunsing yaitu melakukan perbanyakan sekuen. Perbedaanya adalah manik-manik sekuen ditempatkan pada fase padat sehingga mempunyai sensitas yang lebih besar. Selanjutnya probe dinukleotida yang berlabel flulosense dimasukkan ke DNA template. Hasil fluoresensi ditangkap dan direkam untuk menentukan urutan basa. Mekanisme sekuensing SOLiD terlihat pada Gambar 7 (Voelkerdlng 2009).
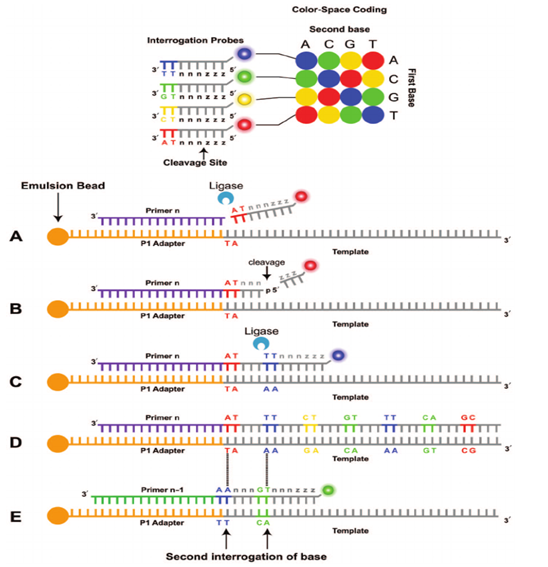
Gambar 7 Proses SOLiD sekuensing. (A) Adaptor P1 dengan primer menempel ke template DNA, (B) Fluoresensi dicatat, (C) Annealing dan ligasi probe berikutnya. (D) Ekstensi lengkap dari primer (n) dan (E) Produk yang diperpanjang dari primer (n) didenaturasi dari adaptor/template.
DAFTAR PUSTAKA
Ansorge W. 2009. Next generation DNA sequencing technigues. Elsevier. 25(4): 195-204
Dhanapal AP. 2012. Genomics of crop plant genetic resources. Adv. Biosci. Biotechnol. 3: 378–385.
Fakruddin, Mazumdar R, Chowdgury A, Hossain N, Mahajan S dan Islam S. 2013. Pyrosequencing a next generation sequencing technologi. World Applied Sciences Journal. 24(12): 155-1571
Franca LTC, Carrilho dan Kist T. 2002. A review of DNA sequencing techniques. Quaterly Reviews of Biophysics. 35: 169–200.
Kling J. 2005. The search for sequencing through bred. Nat. Biotechnol. 23: 1333–1335.
Maxam AM, W Gilbert. 1977. A new method for sequencing DNA. Proc. Natl. Acad. Sci. 74(2): 560-564.
Paterson AH., Freeling M, Tang H dan Wang X. 2010. Insights from the comparison of plant genome sequences. Annu. Rev. Plant Biol. 61: 349–372.
Sanger F, Nicklen dan Coulson. 1997. DNA sequencing with chain-terminating inhibitors. Proc. Nat. Acad. Sci. 74: 5463–5467.
Van K, Rastogi KH, Kim dan Lee S. 2013. Next-generation sequencing technology for crop improvement. SABRAO J. Breed. Genet. 45(1): 84–99.
Voelkerdlng KV, Dames S dan Durtschi J. 2009. Next generation sequensing: from basix research to diagnostic. Clinical Chemistry. 55(4): 641-658.


Leave a comment